今回はAI創薬の第2弾ということで、化合物の符号化(フィンガープリント)をやっていこうと思います!
vol.1を見ていない方は、下のリンクから見れますので是非ご覧ください。
ちなみに、統計検定の記事なども書いてますので、興味のある方は是非そちらも見てもらえたらうれしいです!
前回の記事では、AI創薬がどのようなタスクかというところから、DeepPurposeというライブラリを使ってDavisやKIBAといったデータセットの中身を見てみました。
今回は、化合物の符号化をやっていきます。

はじめに
前回の復習として、ライブラリのインストールと、データセットの中身を見るところまでやっていきましょう。今回はKIBAのみでやっていきます。詳しい説明は前回の記事をご覧ください。
!pip install git+https://github.com/bp-kelley/descriptastorus
!pip install DeepPurposeimport DeepPurpose as DP
from DeepPurpose import dataset
from DeepPurpose import utils
import pandas as pd
X_drugs_K, X_targets_K, y_K = dataset.load_process_KIBA('./data/', binary = False, threshold = 9)df_KIBA = pd.DataFrame({'drugs': X_drugs_K, 'targets': X_targets_K, 'y': y_K})
df_KIBA.head()これらを実行すると、下のような出力となります。
| drugs | targets | y | |
|---|---|---|---|
| 0 | COC1=C(C=C2C(=C1)CCN=C2C3=CC(=C(C=C3)Cl)Cl)Cl | MTVKTEAAKGTLTYSRMRGMVAILIAFMKQRRMGLNDFIQKIANNS… | 11.1 |
| 1 | COC1=C(C=C2C(=C1)CCN=C2C3=CC(=C(C=C3)Cl)Cl)Cl | MSWSPSLTTQTCGAWEMKERLGTGGFGNVIRWHNQETGEQIAIKQC… | 11.1 |
| 2 | COC1=C(C=C2C(=C1)CCN=C2C3=CC(=C(C=C3)Cl)Cl)Cl | MERPPGLRPGAGGPWEMRERLGTGGFGNVCLYQHRELDLKIAIKSC… | 11.1 |
| 3 | COC1=C(C=C2C(=C1)CCN=C2C3=CC(=C(C=C3)Cl)Cl)Cl | MRPSGTAGAALLALLAALCPASRALEEKKVCQGTSNKLTQLGTFED… | 11.1 |
| 4 | COC1=C(C=C2C(=C1)CCN=C2C3=CC(=C(C=C3)Cl)Cl)Cl | MELAALCRWGLLLALLPPGAASTQVCTGTDMKLRLPASPETHLDML… | 11.1 |
データセットを取得することはできましたが、このままでは、モデルに学習させることはできません。
化合物とタンパク質の文字列をコンピュータが処理できる形、つまり何らかの数値ベクトルに変換する必要があります。
ライブラリを使えば、このままの入力でも中身でいい感じにしてくれます。しかし、ゆくゆくは自分でモデルを作りたいとなれば、中でどのような変換が起きているのかを知っておく必要があると思います。
そこで、今回の記事では化合物をフィンガープリント形式に符号化していく方法を解説していきます。
化合物のグラフへの変換やタンパク質については、別の記事で取り上げていこうと思います。
フィンガープリントってなに?
フィンガープリントとは、簡単に言えば、ある化合物中に特定の部分構造が含まれるかを 「0 or 1」のビットで表したものです。
フィンガープリントとは元々指紋という意味ですが、一人一人指紋が異なるように、異なる化合物間ではフィンガープリントは異なります。しかし、類似した化合物では似たものになるように設計されています。これが重要です!
化合物を、それぞれ固有の特徴を表す数値に変換できるのです!
化合物をフィンガープリント表現に変換してみよう!
化合物はデータの中身を見ればわかりますが、
| COC1=C(C=C2C(=C1)CCN=C2C3=CC(=C(C=C3)Cl)Cl)Cl |
上のようなsmiles表現となっています。これをフィンガープリント表現にしていきます。
RDKitというライブラリを使っていきます。RDKitはケモインフォマティクスのためのオープンソースのツールキットであり、化合物をコンピュータ上で扱うためには欠かせないライブラリとなっています。
まずはインストールしていきましょう。
!pip install rdkit次に、必要なライブラリ等をインポートしていきます。
from rdkit import Chem
from rdkit import DataStructs
from rdkit.Chem.Fingerprints import FingerprintMols
from rdkit import rdBase
from rdkit.Chem import AllChem, Draw
import numpy as npRDKitでは、化合物をMolオブジェクトとして扱われます。なので、KIBAデータセットの化合物をまとめてMolオブジェクトに変換しておきます。そして、最初と99999番目の行の化合物を、それぞれmol1とmol2としておきます。
X_drugs_K_mol = [Chem.MolFromSmiles(X_drugs_K[i]) for i in range(len(X_drugs_K))]
mol1 = X_drugs_K_mol[0]
mol2 = X_drugs_K_mol[100000]Molオブジェクトを可視化してみます。左がmol1で右がmol2です。
Draw.MolsToGridImage([mol1, mol2])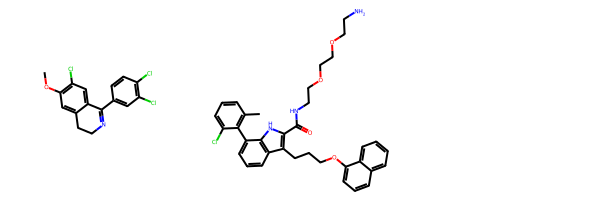
次にフィンガープリントに変換していきます。ここでは詳細は説明しませんが、ECFP4に相当する半径2のモルガンフィンガープリントを使用します。bit数はデフォルト通りに2048にしておきます。
流れとしては、まず、GetMorganFingerprintAsBitVectでフィンガープリントベクトルを生成します。次に、このままでは扱いづらいのでnumpy配列にするために、bit情報を格納する空のnumpy行列を作っておきます。最後に、ConvertToNumpyArrayに、フィンガープリントベクトルと空のnumpy配列を渡すと、numpy配列にbit情報が格納されます。
MORGAN_RADIUS = 2
MORGAN_NUM_BITS = 2048
features_vec = AllChem.GetMorganFingerprintAsBitVect(mol1, MORGAN_RADIUS, nBits=MORGAN_NUM_BITS)
features = np.zeros((0,))
DataStructs.ConvertToNumpyArray(features_vec, features)bit情報が格納されたfeaturesを見てみましょう。
print(len(features))
# -> 2048
print(set(features))
# -> {0.0, 1.0}2048個のビットが0か1に特徴づけされています。つまり、smiles表現だった化合物が、2048個の部分構造があるかないかの2048次元の2値ベクトルに変換されました。
しかし、一つの化合物で同じ部分構造が2カ所以上出てくることもあります。そこで、GetMorganFingerprintAsBitVectの代わりにGetHashedMorganFingerprintを使います。
MORGAN_RADIUS = 2
MORGAN_NUM_BITS = 2048
features_vec = AllChem.GetHashedMorganFingerprint(mol1, MORGAN_RADIUS, nBits=MORGAN_NUM_BITS)
features = np.zeros((0,))
DataStructs.ConvertToNumpyArray(features_vec, features)すると、以下のようになります。
print(len(features))
# -> 2048
print(set(features))
# -> {0.0, 1.0, 2.0, 3.0, 5.0, 8.0}0か1ではなく、2,3,5,8が出てきており、部分構造の出現回数を特徴量にすることができました。
フィンガープリントの可視化
フィンガープリント表現のベクトルにできたことはわかりましたが、正直このままではイメージがつきにくいと思います。そこで、実際にどのような部分構造を捉えているのか可視化してみようと思います。
### Morganフィンガープリント
bitI_morgan = {}
fps_morgan = AllChem.GetMorganFingerprintAsBitVect(mol1, 2, bitInfo=bitI_morgan)先ほどのGetMorganFingerprintAsBitVect関数のbitInfoという引数に、空のディクショナリを渡します。
すると、「bitinfo引数を介して、Morganフィンガープリントの特定のビットに寄与する原子についての情報を手に入れることができます。 この辞書型のデータには、フィンガープリントのビットセットごとに一つのエントリーが取り込まれており、 キー(key)はビットID、値(value)は(アトムインデックス、半径)のタプルのリストとなっています。」(RDKit日本語ドキュメントより引用)
print(bitI_morgan)
# ->{80: ((1, 0), (2, 0), (8, 0)), 87: ((9, 2),), 191: ((10, 1), (23, 1)), ........}確認すると、ここで出てくるビットIDが、先ほどのnumpy配列の0でないビットIDと一致しないのですが、僕自身も理由がよくわかってないので、わかる方がいれば教えてほしいです。
あっているか分かりませんが、特定のビットに寄与した原子があることと、そのビットの部分構造が化合物に実際に含まれていることは別の話、ということで理解しています。
横道にそれましたが、実際に80のビットを可視化してみましょう。以下のコードで可視化できます。
Draw.DrawMorganBit(mol1, 80, bitI_morgan)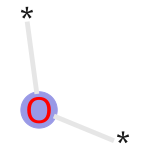
2つ以上でも、タプルにまとめることで可視化できます。以下はmol1のビットIDの小さい方から4番目と5番目を可視化しています。なぜか可視化できないビットがあり、この2つにしました。
理由が分かる方、教えてほしいです、、
morgan_tuples = ((mol1, bit, bitI_morgan) for bit in list(bitI_morgan.keys())[4:6])
Draw.DrawMorganBits(morgan_tuples)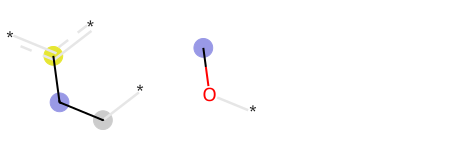
ちなみに、ビット情報の中心元素が青色の丸、芳香族元素が黄色の丸らしいです。

さいごに
今回は、化合物をフィンガープリントに変換して、さらにそれをnumpy配列にしてみました。これによって、機械学習モデルに入力できる形になりました。
これ以外にもいろいろな符号化方法があります。次はグラフ表現をしてみたいと思います。



コメント