この記事では、AI創薬のためのデータセットを扱ってみようと思います!
そもそもAI創薬ってなに?
そもそも、AI創薬とはどのようなタスクでしょうか
広い意味では、創薬のいろいろなフェーズにAIを活用するということになります。
ここでは、特に、薬物候補となる化合物をスクリーニングする段階でAIを活用することを考えます。
スクリーニングの段階でAIを活用する際には、化合物とターゲットとなるタンパク質が結合するかどうかをAIで予測するというタスクを考えます。
つまり、化合物とタンパク質をコンピュータの処理できる形で表し、AIによってその関係性を予測するタスクです。
関係性は、結合するかしないかの二値分類や、IC50,EC50,Ki,Kdといったアフィニティ(結合のしやすさ)の数値などで表されます。
以下の図を見ると分かりやすいと思います。
化合物は文字列やフィンガープリント、グラフで表現し、タンパク質は文字列やグラフなどで表現します。これにより、コンピュータで処理できる形になりました。そして、決定木などの古典的解学習手法や、RNN、CNNといったディープラーニング手法などでアフィニティを予測していくことになります。
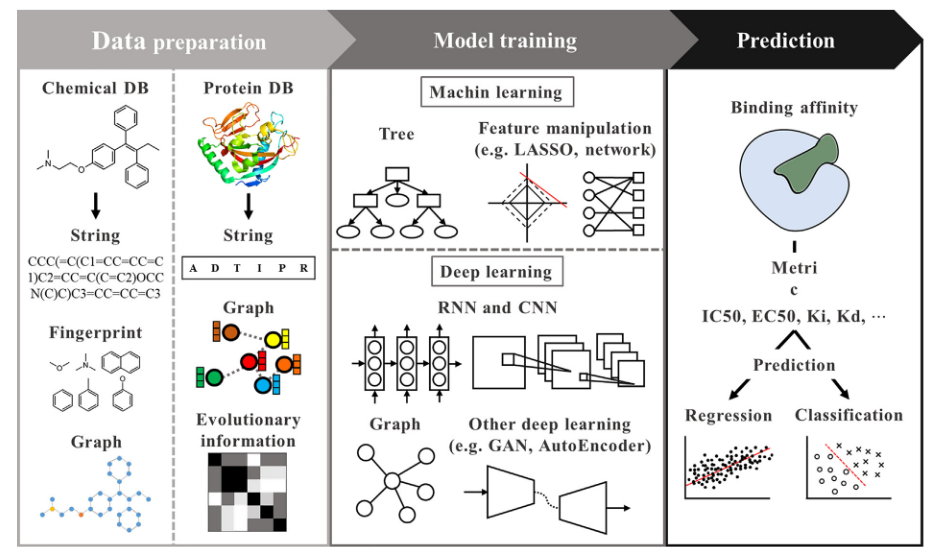
最近では、データの蓄積やディープラーニングの発展に伴い、AI創薬の分野も盛り上がっています。
データセットを見てみよう!
では、AI創薬がどんな感じか分かったところで、さっそくAI創薬に使われるデータセットを見てみましょう!
多くのデータセットがありますが、ここではDavisデータセットとKIBAデータセットを見てみようと思います。下の表のハイライトを掛けている二つです。二つとも比較的小さいデータセットです。
compoundsが化合物、proteinsがタンパク質、interactionsがcompound-proteinの相互作用の情報です!
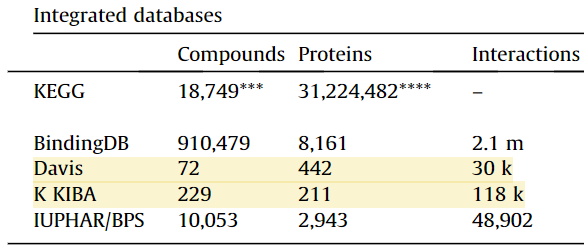
それでは、実際にこれらのデータセットの中身を見てみましょう。
ここではDeepPurposeというライブラリを使っていきます。

DeepPurposeはDTI(drug target interactions)予測のためのデータセットやモデルを数多くサポートしており、簡単なコードでこれらのツールを使うことができます!
まずはインストールしましょう。以下を実行するとDeepPurposeが使えます。下の行だけでは動かないので注意が必要です。
!pip install git+https://github.com/bp-kelley/descriptastorus
!pip install DeepPurpose次に、必要なライブラリをインポートしていきます。
import DeepPurpose as DP
from DeepPurpose import dataset
from DeepPurpose import utils
import pandas as pdまず、KIBAデータセットを取得してみます。なんと次の一行で取得できます!最初の引数直下にデータが格納されます。なので、ここでは ./data/ 直下にデータが格納されます。
X_drugs_K, X_targets_K, y_K = dataset.load_process_KIBA('./data/', binary = False, threshold = 9)このままではどのようなデータか見にくいので、pandasを使ってデータフレームにしてみましょう。
df_KIBA = pd.DataFrame({'drugs': X_drugs_K, 'targets': X_targets_K, 'y': y_K})
df_KIBA.head()出力は下の表のようになります。
| drugs | targets | y | |
|---|---|---|---|
| 0 | COC1=C(C=C2C(=C1)CCN=C2C3=CC(=C(C=C3)Cl)Cl)Cl | MTVKTEAAKGTLTYSRMRGMVAILIAFMKQRRMGLNDFIQKIANNS… | 11.1 |
| 1 | COC1=C(C=C2C(=C1)CCN=C2C3=CC(=C(C=C3)Cl)Cl)Cl | MSWSPSLTTQTCGAWEMKERLGTGGFGNVIRWHNQETGEQIAIKQC… | 11.1 |
| 2 | COC1=C(C=C2C(=C1)CCN=C2C3=CC(=C(C=C3)Cl)Cl)Cl | MERPPGLRPGAGGPWEMRERLGTGGFGNVCLYQHRELDLKIAIKSC… | 11.1 |
| 3 | COC1=C(C=C2C(=C1)CCN=C2C3=CC(=C(C=C3)Cl)Cl)Cl | MRPSGTAGAALLALLAALCPASRALEEKKVCQGTSNKLTQLGTFED… | 11.1 |
| 4 | COC1=C(C=C2C(=C1)CCN=C2C3=CC(=C(C=C3)Cl)Cl)Cl | MELAALCRWGLLLALLPPGAASTQVCTGTDMKLRLPASPETHLDML… | 11.1 |
drugsはsmiles表現(Simplified Molecular-Input Line-Entry System)、targetsはアミノ酸配列の文字列にエンコードされてます。
次にDavisデータセットを取得してみましょう。同じように一行で書けます!
X_drugs_D, X_targets_D, y_D = dataset.load_process_DAVIS('./data/', binary = False, convert_to_log = True, threshold = 30)KIBAと同じように、データフレームにして可視化してみましょう。
df_DAVIS = pd.DataFrame({'drugs': X_drugs_D, 'targets': X_targets_D, 'y': y_D})
df_DAVIS.head()出力は下の表のようになります。
| drugs | targets | y | |
|---|---|---|---|
| 0 | CC1=C2C=C(C=CC2=NN1)C3=CC(=CN=C3)OCC(CC4=CC=CC… | MKKFFDSRREQGGSGLGSGSSGGGGSTSGLGSGYIGRVFGIGRQQV… | 7.366532 |
| 1 | CC1=C2C=C(C=CC2=NN1)C3=CC(=CN=C3)OCC(CC4=CC=CC… | PFWKILNPLLERGTYYYFMGQQPGKVLGDQRRPSLPALHFIKGAGK… | 5.000000 |
| 2 | CC1=C2C=C(C=CC2=NN1)C3=CC(=CN=C3)OCC(CC4=CC=CC… | PFWKILNPLLERGTYYYFMGQQPGKVLGDQRRPSLPALHFIKGAGK… | 5.000000 |
| 3 | CC1=C2C=C(C=CC2=NN1)C3=CC(=CN=C3)OCC(CC4=CC=CC… | PFWKILNPLLERGTYYYFMGQQPGKVLGDQRRPSLPALHFIKGAGK… | 5.000000 |
| 4 | CC1=C2C=C(C=CC2=NN1)C3=CC(=CN=C3)OCC(CC4=CC=CC… | PFWKILNPLLERGTYYYFMGQQPGKVLGDQRRPSLPALHFIKGAGK… | 5.000000 |
KIBAと同じような表現となっていることが確認できました!
さいごに
今回はDeepPurposeを使って、DavisとKIBAというAI創薬で使われるデータセットを可視化してみました。
次回はRDkitというライブラリを使って、取得したこれらのデータを色々な形式に変換していこうと思います!


コメント